

近日,我室刘宗保老师在国际学术期刊《Journal of Environmental Management》发表了一篇题为“Deep sequencing reveals comprehensive insight into the prevalence, mobility, and hosts of antibiotic resistance genes in mangrove ecosystems”的研究论文,珍稀濒危动植物生态与环境保护教育部重点实验室为该论文第一单位,刘宗保老师为第一作者,深圳大学李猛教授为通讯作者。《Journal of Environmental Management》为中科院大类学科一区TOP期刊,影响因子8.91。

图1 论文摘要图
滨海红树林因经常受到水产养殖废水和城市污水污染,是潜在的抗生素抗性基因(Antibiotic resistance gene,ARG)储存库。先前针对红树林生态系统中ARG的研究大多是基于qPCR和基于短序列分析的宏基因组方法,很难全面鉴别它们的遗传背景及宿主信息。该研究利用宏基因组组装和分箱方法,通过超高深度测序(>110 Gb/样)全面揭示了ARG在我国东南沿海6个红树林生态系统中的分布、潜在移动性和宿主。结果显示,在红树林生态系统中共发现了348种ARG,涉及26类抗生素。基于网络共现性分析,其中6种ARG基因亚型可作为红树林生态系统中其他100种ARG的丰度指示。此外,在检测出的348种ARG中,有250种被注释为与可移动基因元件相关,这表明这些红树林生态系统中的ARG具有较高的扩散风险。最后,通过对超过6,000个基因组草图中ARG的鉴别查找,发现共有42个细菌门类被确定为ARG的潜在宿主。其中,21.97%的宿主是潜在多重耐药菌株,包括人类和动物的条件性致病菌。
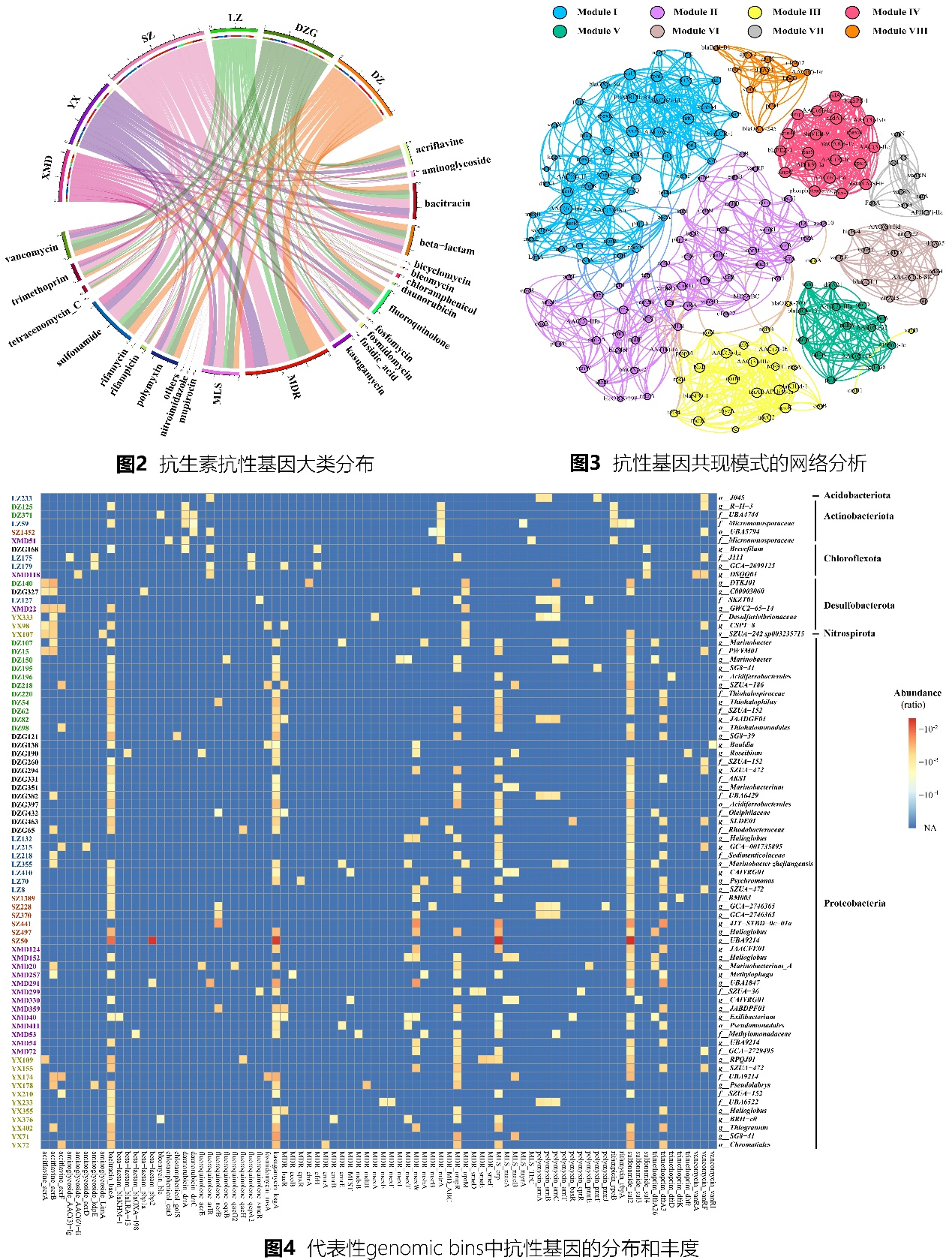
这项研究极大地扩展了我们对红树林生态系统中抗生素抗性基因分布和传播的认识,为抗生素耐药性的风险评估和管理提供了重要依据。该研究受到了国家自然科学基金、广西科技基地和人才专项等项目的支持。
论文链接:https://doi.org/10.1016/j.jenvman.2023.117580